

전 세계적으로 5만 개가 넘는 인간 관련 유전질환이 있는데 대부분 게놈의 점 돌연변이가 원인이다.희귀병의 경우 80%가 유전자 결함으로 발생했고,이들 유전자 결함 중 점 돌연변이가 60%를 차지했다.예를 들어 다발성 신경섬유종(Neurofibromatosis)의 경우 Neurofibromin 1/2 유전자의 염기 돌연변이에 의해 발생한다.게놈 점 돌연변이에 대한 연구는 중요한 생물학적 원리뿐만 아니라 인간의 질병 치료 수단 개발에도 도움이 될 것으로 보인다.
CRISPR-Pro 시스템은 게놈 편집의 마술 메스로 꼽힌다.특히 CRISPR-Pro 시스템과 세포 상동재조합 복구 메커니즘 (HDR) 을 기반으로 한 유전자 편집 기술은 유전자점 돌연변이 유전병 방향에 큰 응용 잠재력을 보이고 있다.그러나 상동재조합의 효율성이 떨어지고,비 상동 재조합 메커니즘이 작동하기 쉬운 예측불허의 돌연변이 유형 등의 문제가 있어 보다 간편하고 효율적이고 정교한 기술 개발이 요구되고 있다.단일 염기 편집 시스템은 이런 배경에서 개발됐다.
단일염기편집시스템에는 시이토신(Cytosine)염기편집기(CBE),아데닌(Adenine)염기편집기(ABE),구아닌(Guanine)편집기(GBE),Prime Editor가 있다.이들 편집기는 DNA 이중사슬 절단결핍(DSB)이 생기지 않아 세포의 이중 요소 파손(double-strand breakage) 복원 메커니즘을 촉발하지 않는다.상기 네 가지 편집기를 각각 소개한다.
사이토신 편집기
시이토신(Cytosine) 편집기(CBE)는 CRISPR/Cas9 시스템을 기반으로 개발되었으며,sgRNA와 융합단백질 2가지로 구성되어 있다.Cas9 핵산효소의 RucC 와 HNH 의 두 기능 영역을 돌연변이하여 돌연변이형 dCas9(dead Cas9) 또는 nCas9(nickase nCas9) 를 발생시킨 다음 돌연변이형 Cas9,사이토신 탈아미노효소,우라실(Uracil) glycosylase 억제자를 융합시켜 융합단백질을 형성한다.시이토신 편집기의 작동원리는 sgRNA의 안내를 받아 특정 유전체 위치에 단백질을 융합한 뒤 시이토신 탈아미노효소가 C를 U로 바꾼 뒤 U가 DNA 복제 과정에서 T로 바뀌는 것이다(그림1).이 과정에서 우라실(Uracil) glycosylase 억제자는 중간체 U의 절제를 억제하고 T의 전환율을 높이는 역할을 한다.
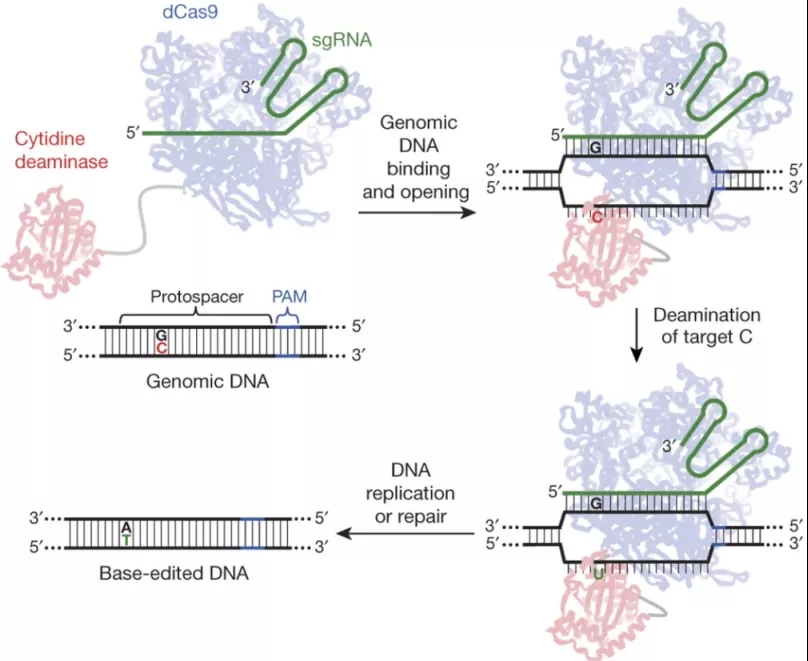
그림 1. CBE의 작동 원리 [1]
1세대 시이토신 편집기 CBE1(rAPOBEC1-XTEN-dCas9)에서는 융합단백질이 Rat에서 유래한 rAPOBEC1(Rat 사이토신 탈아미노효소Cytosine deaminase)와 dCas9로 구성되어 있다.편집 활성 창은 약 5개의 염기이고 표적 위치는 4-8번째 염기이다.CBE1은 체외에서는 편집효율이 높지만 포유동물에서는 편집효율이 매우 낮다.연구진 분석 결과 DNA 글리코실라제(Uracil DNA glycosylase,UDG)가 중간체인 UG를 인식해 염기절제복구경로(BER)를 통해 CG로 다시 복원했기 때문으로 보인다.따라서 염기 절제 복원 경로를 억제해 시이토신 편집기의 편집 효율을 높일 수 있다.그리고 연구자들은 rAPOBEC1-XTEN-dCas9에 Uracil DNA glycosylase(UGI)를 융합하여 2세대 시이토신 편집기 CBE2(raPOBEC1-XTEN-dCas9-UGI)를 개발했다[1].CBE2는 BE1보다 3배 높은 최대 20%까지 높은 편집 효율성을 자랑한다.실천 과정에서 CBE1과 CBE2는 DSB를 만들지 않기 때문에 게놈에 비표적염기 삽입과 부재,교체가 발생할 확률이 매우 낮다.
그러나 연구자들은 여전히 편집 효율을 높이고자 dCas9 를 nCas9(Cas9 nickase nCas9 D10A) 로 교체하고 3 세대 시이토신 편집기 CBE3 (rAPOBEC-XTEN-nCas9(D10A)-UGI) 을 구축했다.nCas9 는 편집되지 않은 DNA 체인 (G 가 있는 DNA 체인) 을 절단하는 활성으로 DNA 단일 체인 틈새를 형성한다.그리고 세포는 염기가 잘못 짝짓기를 하는 복원경로(MMR)가 작동하고 편집 체인 (편집 후 u 가 있는 DNA 체인) 을 템플릿으로 복구한다 (그림 2).CBE3 은 CBE2 보다 2-6 배 높은 편집 효율성을 가지고 있지만,CBE3 은 C 를 T 로 변환하는 것 외에도 C 를 G 또는 A 로 변환할 수 있으며 비상동성(homology) 복구 메커니즘(NHEJ)인 Indel을 유발할 수 있다.
편집 효율성을 더욱 높이기 위해 개발자는 CBE3를 기반으로 두 번째 카피의 UGI를 융합하는 한편,rAPOBEC1,nCas9과 UGI 사이의 이음 길이를 최적화하여 4세대 시이토신 편집기 CBE4(rAPOBEC-XTEN-nCas9(D10A)-2UGI)를 구축하였다[2].CBE4는 CBE3에 비해 C-G와 C-A 유전자 편집 생성물을 2.3배 줄였다.또한 CBE4에 더해 Phage Mu에서 유래한 Gam단백질을 융합하거나 다른 수의 (NLS,Nuclear localization sequence)핵위치 서열를 추가하고 회사별로 최적화된 암호부서열을 사용하는 등의 방법으로 NHEJ 복구 경로 발생을 줄이고 편집 효율성을 향상시켰다.
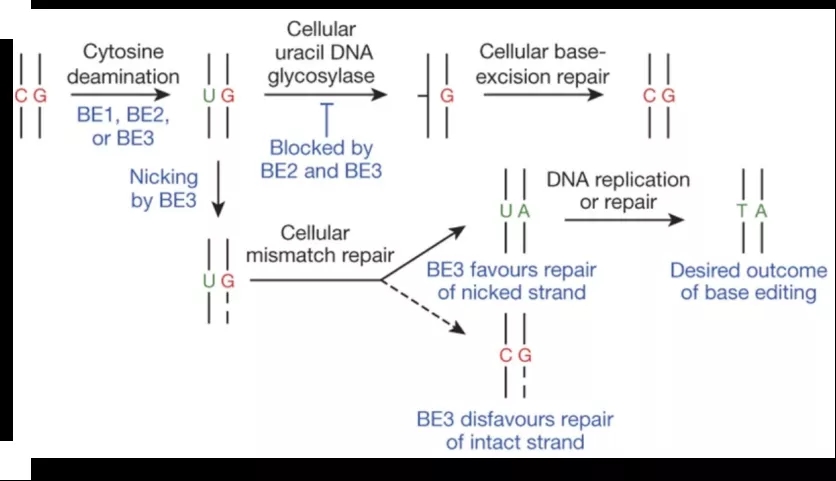
그림 2. CBE의 업그레이드 [1]
아데닌 편집기(Adenine)
아데닌 편집기(ABE)는 CBE와 유사하게 ABE의 융합단백질은 nCas9(D10A)과 인위적으로 변형된 아데닌 탈아미노효소(deaminase)로 이루어져 있으며 알킬아데닌 DNA glycosylase(AAG)의 활성을 억제하지 않고 ABE7.10(ecTadA-ecTadA*-nCas9)를 만들어낸다[3].그 작동 원리는 sgRNA 의 안내에 따라 융합 단백질을 과녁 시퀀스 영역에 결합하고 일정 영역의 아데닌(A) deamination creatinine(I)을 만들고 I를 G로 바꿔 A에서 G로 변환한다(그림3). ABE의 편집 효율을 더욱 높이기 위해,연구자들은 ABE7.10을 기반으로 NLS를 융합하고,회사별로 최적화된 암호 서브시퀀스를 사용하는 등의 방법으로 A에서 G로의 변환 효율을 향상시켰다[4].
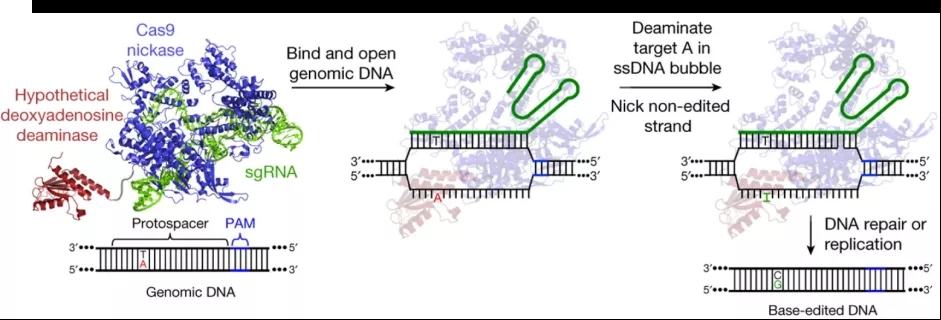
그림 3. ABE의 작동 원리 [3]
GBE
GBE 기술은 CBE를 기반으로 발전했다.CBE는 C를 U로 바꾼 뒤 몸속의 UNG 효소가 U염기를 제거하기 때문에 퓨린 피리미딘 없는 AP 상태(apurinic/apyrimidinic)를 형성한다.AP상태에서는 염기절제 복구 메커니즘을 촉발하여 U를 다시 C로 바꿀 수 있지만 다른 세 가지 염기로 복구할 수 있다[5].CBE는 UNG의 활성을 억제함으로써 C에서 U에서 T로의 변환 효율을 높이는 것이다.반면 GBE는 AP 상태를 이용해 다른 염기로의 전환이 가능하다.연구자들은 UNG와 nCas9을 융합해 대장균에서 C를 A로 변환하는 것이 주요 편집 결과임을 알아냈다.그러나 포유동물 세포인 HEK293T에서는 C가 G로 바뀌는 것을 주요 편집 결과가 있다[6](그림4).AP상태에서 세포에는 AP상태를 다른 염기로 복원하는 미지의 복구 메커니즘이 존재함을 알 수 있다.따라서 이 기술은 계속 보완되어야 한다.
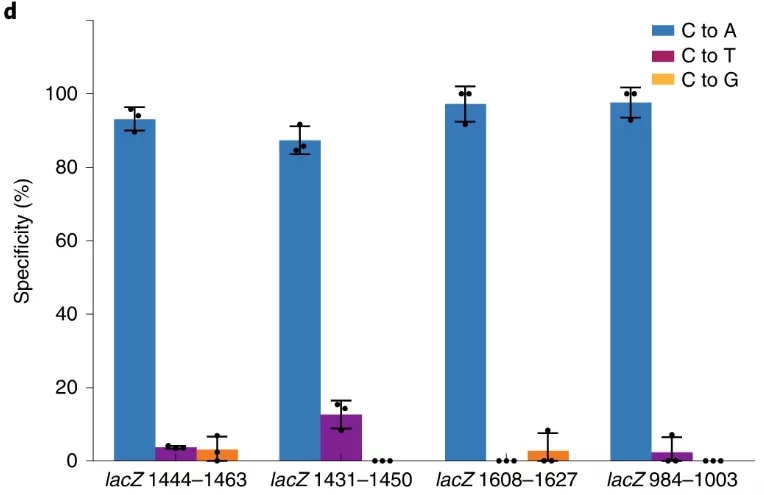
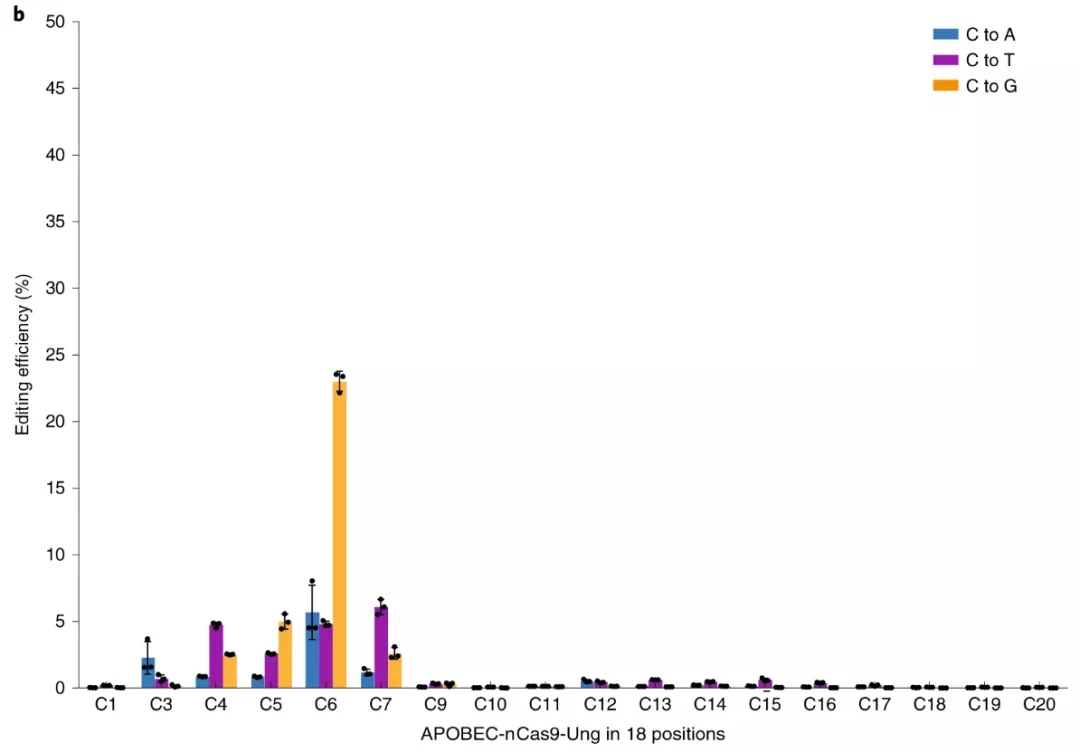
그림 4. 대장균 또는 HEK293T에서 GBE의 편집효과 [6]
(A) 대장균에서의 GBE 편집 효과.(B) HEK293T에서의 GBE 편집 효과
선도편집기
CBE와 ABE는 C에서 T와 A에서 G로의 전환이 가능하지만 다른 서로 다른 염기 간의 전환은 여전히 제한적이어서 연구자들은 모든 염기전환에 적응할 수 있는 기술인 선도편집기(Prime Editor,PE)를 계속 개발해왔다.PE는 염기의 교체뿐만 아니라 다염기의 정확한 삽입 및 제거를 효과적으로 수행할 수 있다[7].PE 역시 CRISPR/Cas9 시스템을 기반으로 개조된 sgRNA(pegRNA)와 융합단백질로 구성되어 있다.PegRNA 는 원래 sgRNA 를 기반으로 상보적으로 부러질 수 있는 표적 DNA 체인 3' 의 끝을 추가하는 시퀀스 (PSB) 와 돌연변이 지점이 있거나 결여된 목적 서열(RT 템플릿)을 삽입하다.융합단백질은 nCas9(H840A)과 역전사 효소가 융합해 만들어진다.PE 의 작동 원리는 pegRNA 가 융합 단백질을 표적 서열 영역으로 유도하고,nCas9은 편집된 DNA를 끊어내고 이어 PSB 는 끊어진 과녁 DNA 를 결합해 역전사 효소와 RT 템플릿 아래 돌연변이 부위를 포함하는 목적 서열을 역전시켰다.반응이 끝나면 표적 돌연변이가 있는 3'flap 구조의 DNA 사슬과 아무런 돌연변이가 없는 5'flap 구조의 DNA 사슬이 형성된다.세포 내 5'flap 구조는 구조특이성 내체효소에 의해 쉽게 인식되고 제거되며,이후 DNA 연결 및 복구를 통해 정확한 유전자 편집을 가능하게 된다 (그림 4).
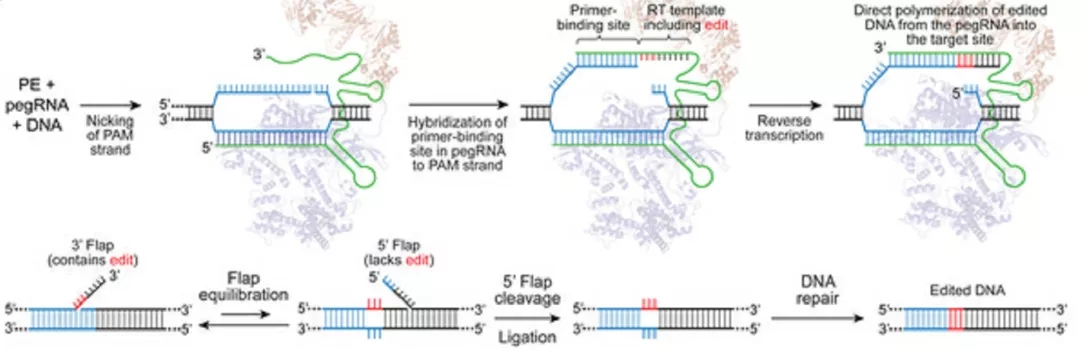
그림 5. PE의 작업 원리 [8]
결론
현재 CRISPR-Pro 시스템과 세포상동성(homologous) 재조합 복구 메커니즘에 기초한 유전자 편집 기술의 발전이 비교적 성숙되었다.CRISPR-Pro 시스템을 통해 특정 유전체 위치에 이중 사슬 DNA 결핍이 발생하여 세포의 상동(homologous) 재조합 복원 메커니즘을 작동시켜 목적의 돌연변이가 포함된 고도의 상동(homologous)DNA 서열을 복원함으로써 염기전환의 목적을 달성한다.이 기술은 이미 유전자 치료와 유전자 기능 연구 등에 활용되고 있다.그러나 일부 단점은 상동(homologous) 재조합의 효율성이 떨어진다든지,NHEJ 복구방식의 작동이 용이하여 예측 불가능한 돌연변이를 초래하는 등의 단점도 있다.단염기편집시스템은 CRISPR/Cas9 시스템에 기반하여 돌연변이 Cas9 핵산내절체효소의 절단활성을 통해 DNA 이중 연쇄 결핍이 생기지 않아 Indel 도입의 위험을 줄인다.아울러 돌연변이형 Cas9에 탈아미노효소(deaminase) 등 다른 기능단백질을 융합해 염기를 달리하는 변환도 이뤄진다.단순성,효율성,Indel 도입 쉽지 않은 특징은 유전자 치료 분야에서 중요하게 여겨지기 때문에 단일 염기 편집 시스템은 유전자 기능 연구와 유전자 치료 등 분야에서 강력한 지속적 동력을 제공할 것이다.
참고 문헌:
[1] Komor, Alexis C., et al. “Programmable Editing of a Target Base in Genomic DNA without Double-Stranded DNA Cleavage.” Nature, vol. 533, no. 7603, 2016, pp. 420–424.
[2] Komor, Alexis C., et al. “Improved Base Excision Repair Inhibition and Bacteriophage Mu Gam Protein Yields C:G-to-T:A Base Editors with Higher Efficiency and Product Purity.” Science Advances, vol. 3, no. 8, 2017.
[3] Gaudelli, Nicole M., et al. “Programmable Base Editing of A•T to G•C in Genomic DNA without DNA Cleavage.” Nature, vol. 551, no. 7681, 2017, pp. 464–471.
[4] Koblan, Luke W., et al. “Improving Cytidine and Adenine Base Editors by Expression Optimization and Ancestral Reconstruction.” Nature Biotechnology, vol. 36, no. 9, 2018, pp. 843–846.
[5] Nishida, Keiji, et al. “Targeted Nucleotide Editing Using Hybrid Prokaryotic and Vertebrate Adaptive Immune Systems.” Science, vol. 353, no. 6305, 2016.
[6] Zhao, Dongdong, et al. “Glycosylase Base Editors Enable C-to-A and C-to-G Base Changes.” Nature Biotechnology, vol. 39, no. 1, 2021, pp. 35–40.
[7] Doman, Jordan L., et al. “Evaluation and Minimization of Cas9-Independent off-Target DNA Editing by Cytosine Base Editors.” Nature Biotechnology, vol. 38, no. 5, 2020, pp. 620–628.
[8] Anzalone, Andrew V., et al. “Search-and-Replace Genome Editing without Double-Strand Breaks or Donor DNA.” Nature, vol. 576, no. 7785, 2019, pp. 149–157.
About Cyagen
Cyagen은 16 년 동안 발전해 전 세계 수만 명의 과학자에게 봉사했으며, 제품과 기술은 CNS(Cell,Nature,Science) 3 대 저널을 포함한 4500 여 편의 학술 논문에 직접 적용되었다.유전자녹아웃,유전자노크인,조건부 녹아웃 모델 맞춤형 서비스 외에도 Cyagen에는 전문적인 수술질환 모델팀이 있어 다양하고 세밀한 작은 동물수술 질병모델을 제공할 수 있다.약물선별평가 마우스 플랫폼은 구미 업계 리더로부터 도입한 면역결핍마우스,심혈관 및 알츠하이머병 등의 연구에 사용되는 인간화 마우스를 제공할 수 있다.국제 표준화 무균쥐 기술 플랫폼은 무균마우스,무균동물 맞춤형 서비스,미생물 균군 이식 서비스 등 무균동물 모델에 기반한 각종 제품과 서비스를 제공할 수 있으며,Cyagen의 성숙하고 안정적인 유전자 편집 마우스 플랫폼과 결합하여 균군과 유전자의 상호작용 메커니즘을 연구하는데 도움을 줄 수 있다.




영업일 기준 1-2일 내에 답변해 드리겠습니다.